生命科學院周信宏/溫進德團隊研究成果登上 Genome Research
突變是演化的原動力,它改變DNA序列 (基因型),進而影響基因功能及生存優勢 (表型)。了解突變的效應和基因型-表型的關聯是生命科學的重要課題,如能獲取這些資訊,預測演化走向及設計人工生命體便非遙不可及的夢想。但要完整掌握基因型-表型的關聯極為困難,因為基因型的總量隨DNA長度呈指數成長,光是10個鹼基組成的DNA片段就有超過一百萬種 (4^10) 基因型 (註:人類基因體含32億個鹼基),傳統的研究方法難以克服此數量障礙。
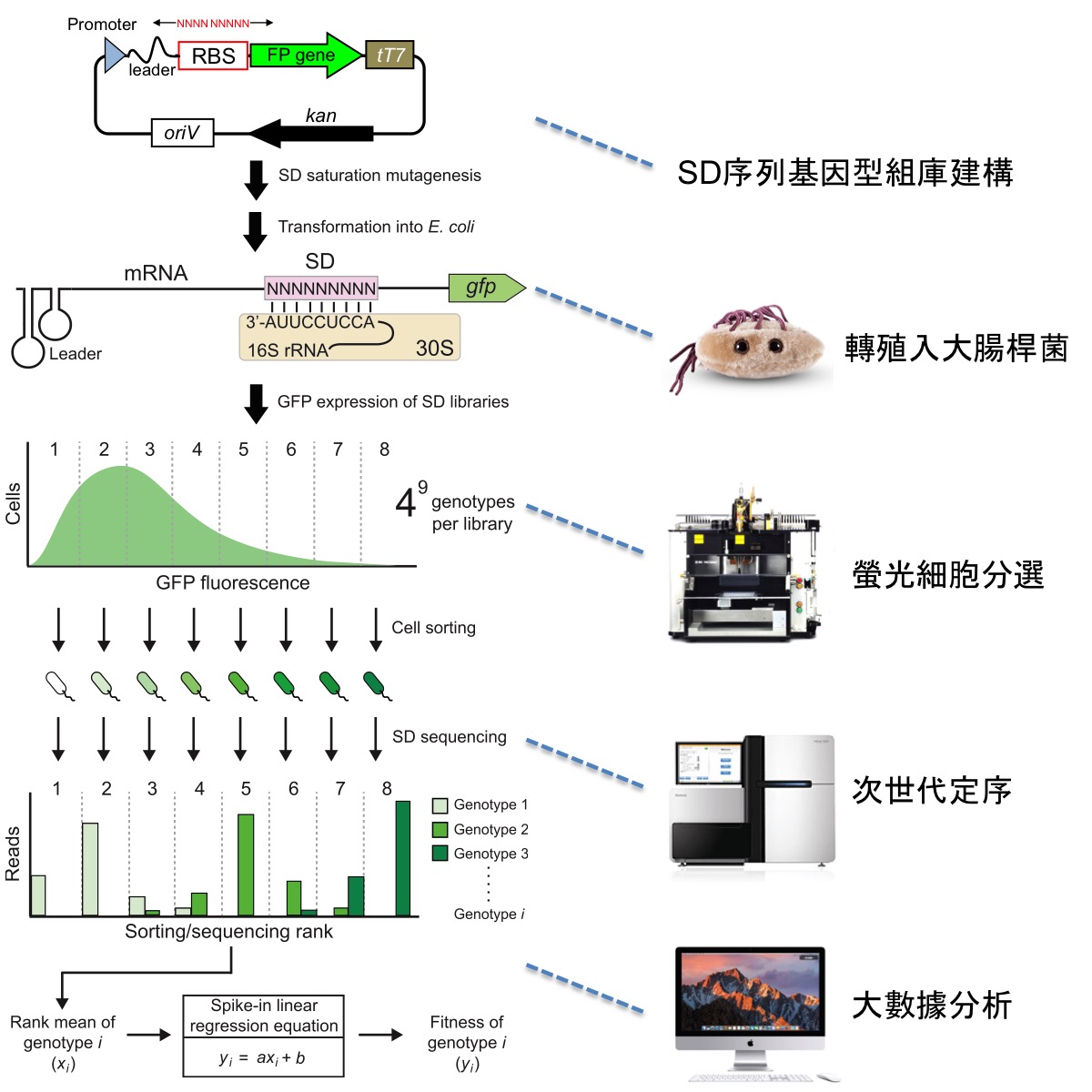
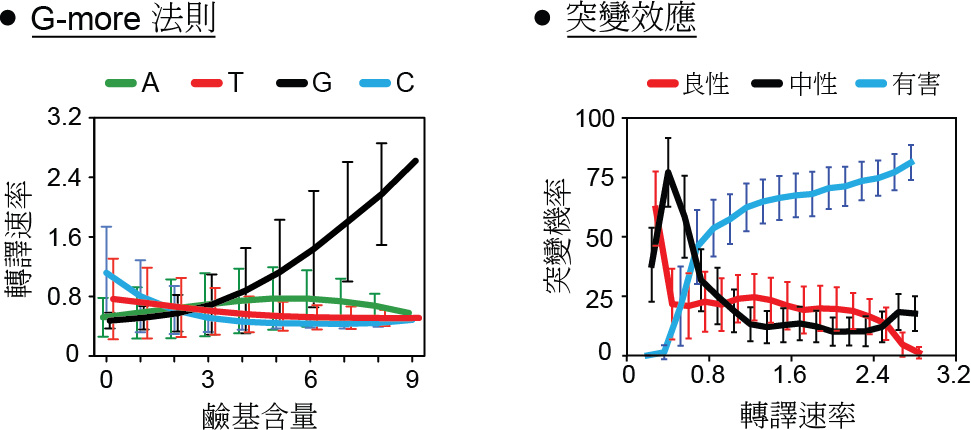
為了揭開基因型-表型關聯的全貌,生科系周信宏老師 (圖一,右三) 發展一套結合螢光細胞分選及次世代定序技術的高通量實驗平台 (圖二),能同時建構百萬種基因型,並觀測每個基因型產生的表型,他以細菌的 SD (Shine-Dalgarno) 序列作為研究題材。SD序列為基因上游9個鹼基構成的區塊,SD序列影響核糖體的附著並決定基因的蛋白轉譯速率,教科書將SD定義為一段富含 A、G鹼基的序列,但自然界SD序列的基因型其實很多元。周老師以高通量技術在四種大腸桿菌基因的上游產生所有種類 (4^9) 的SD基因型,並觀測每個基因型的表型 (轉譯速率)。他發現四種基因上游對SD功能的影響各有不同,增加了SD基因型-表型關聯的複雜性,但透過大數據分析周老師從看似雜亂的表象中找到簡單的規則 (圖三左):G鹼基越多 (G-more) 的SD基因型通常具較高的轉譯速率,反之,A鹼基的多寡則與轉譯速率無關。生細所溫進德老師 (圖一,左一) 進行單分子實驗並驗證了這項觀察,實驗結果顯示G鹼基提高SD序列與核糖體的親和力,進而增加蛋白轉譯速率。G-more法則簡化了人工SD序列的設計,使合成生物學家可更精準的預測並控制基因表現。
另一方面,周老師從演化及遺傳學的角度分析SD基因型-表型關聯的特性,他發現轉譯速率越低的SD基因型獲得良性突變的機率越高,令人意外的是,SD序列在朝向高轉譯速率的基因型演化的過程中,獲得良性突變的機率仍維持一穩定值 (圖三右),此現象與演化理論的預測相左。上述兩種特性都可增進SD序列的演化潛力 (Evolvability),至於兩特性是否為生物系統所共有則有待後續研究進一步探討。
本篇的共同第一作者為生科系大學部畢業生郭學庭 (圖一,右二) 、電機系大學部畢業生簡瑞霖 (圖一,右一)、及生科系碩班畢業生鄭遠儒。其他作者包含: 生科系碩班畢業生李昀儒以及基因體與系統生物學學位學程博班畢業生陳臆嵐 (圖一,左二)。研究經費來自科技部及臺大學術研究生涯發展研究計畫。
周信宏老師特別感謝臺大提供微生物專用的流式細胞分選儀 (BD FACSJazz; 圖一,左三),歡迎校內有微生物分選需求的團隊洽詢及使用,以發揮此儀器的最大功用。
Genome Research論文全:http://www.genome.org/cgi/doi/10.1101/gr.260182.119
生命科學系 周信宏助理教授:https://choulab.wordpress.com/
分子與細胞生物學研究所 溫進徳副教授:https://jdwen.wordpress.com/