大數據探勘癌症組織的細菌轉錄圖譜
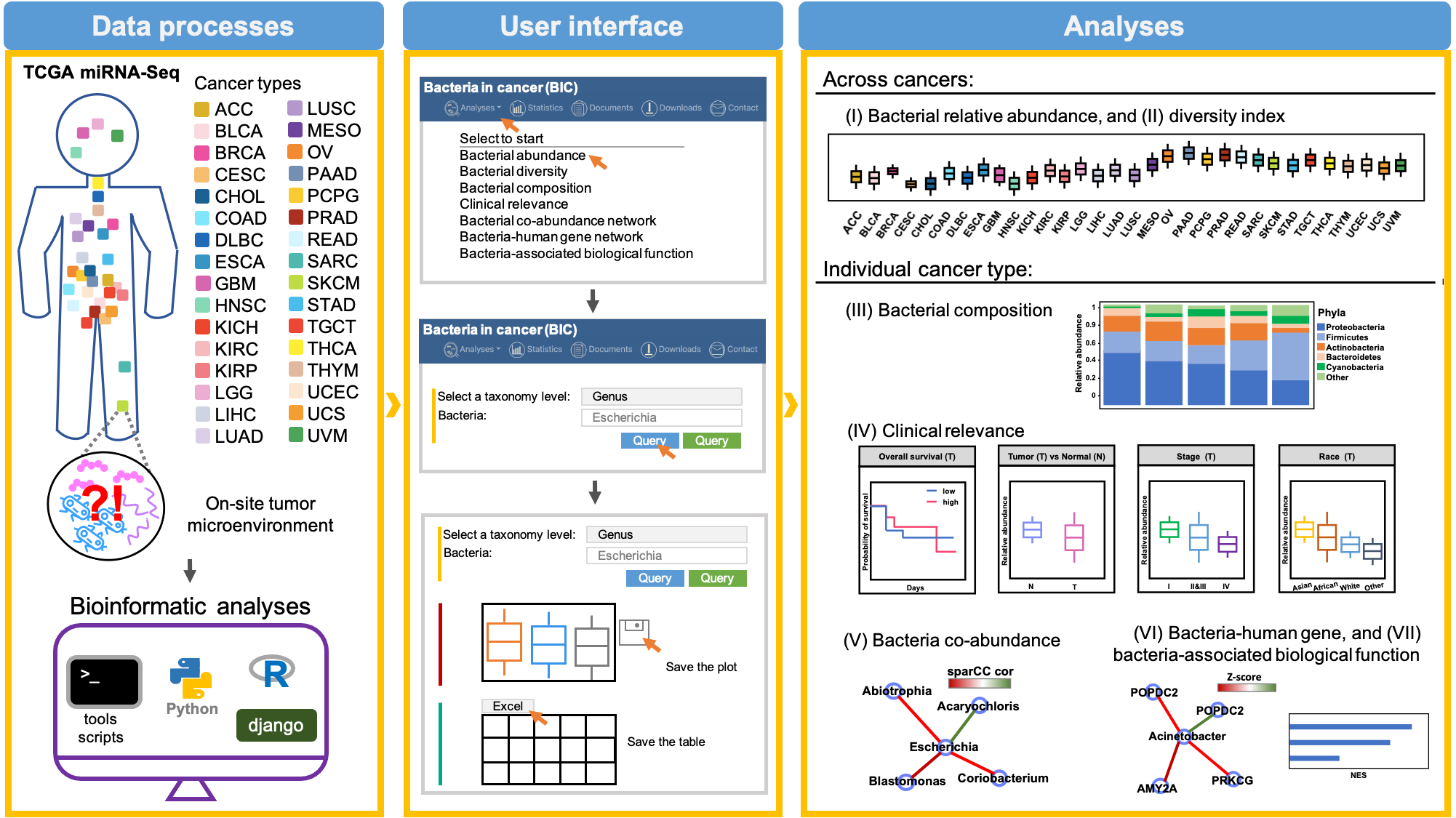
以往認為人類腸道以外的內臟是無菌的,尤其腫瘤組織中偵測到的細菌常被認為是取樣時的汙染。然而,近來越來越多的證據顯示癌症組織中存在各種微生物。本次研究基於「物盡其用」的概念,將癌症基因體圖譜(The Cancer Genome Atlas, TCGA)的人類癌症微小核醣核酸序列資料進行大數據分析,將原本沒有對應到人類基因而該被丟掉的序列再利用,與細菌基因資料進行序列比對,從而獲取癌症組織中存在的細菌種類與表現數量等資訊,進而建構「BIC」資料庫,提供與微生物群落相關的癌症微環境生物資訊。此項以臺大師生為首的跨校團隊,所得的研究成果發表於生物化學研究領域排名第一的期刊《核酸研究》(Nucleic Acids Research)。
BIC資料庫探勘來自32種癌症類型含有1萬362個病人組織樣本的大數據序列資料,運用多項生物資訊分析獲得細菌組成及其豐富度、細菌歧異度、細菌間的共豐度網路、各類細菌的臨床關聯性、相關的人類基因表達以及生物功能等各類資料與分析結果,並提供線上視覺化分析查詢介面,讓使用者能更方便有效的利用與下載這些資訊。BIC資料庫為使用者可自由使用的公開資料庫,且所有的開發程式碼皆公開在GitHub,歡迎生物資訊相關社群的研究人員與愛好者可以此開發其他的應用。
此項研究成果由生命科學系暨生醫電子與資訊學研究所特聘教授,兼計算與系統生物學中心主任阮雪芬,與國立陽明交通大學生物醫學資訊所教授黃宣誠合作的研究團隊,由國科會(原科技部)和臺大深耕計畫資助,以及國家高速網路與計算中心的計算資源下完成。第一作者為生醫電子與資訊學研究所博士生陳凱普,參與研究的團隊成員還包括臺大醫院許家郎博士和生醫電子與資訊學研究所教授歐陽彥正。
陳凱普同學患有罕見疾病脊髓性肌肉萎縮症,能夠順利完成此項研究工作也要特別感謝臺大和生科院於校園中建置無障礙空間,讓陳同學能無憂地在校努力不懈,除了充實知識外,也學習生物資訊與資料庫建構技術,因而有此研究成果產生。