臺大研究成果系列報導—全球農業科技與基因體科學碩士學位學程(Global ATGS)教授伊藤剛將大規模基因體定序數據予以視覺化
現代分子生物學中的全基因體研究
今日所見的分子生物學,已和上個世紀有所不同。雖然大眾已經明白 DNA 中蘊含著關於生物過程的資訊,技術上卻難以將單一細胞中所有 DNA 分子的「數據」予以解碼,這數據就是所謂的基因體(genome)。過去 20 年來,一系列 DNA 定序的技術革新,造成分子生物學歷經大幅度改革,但這些革新其實有一部分始於 1990 年代。所謂「活體內」(in vivo)的生物反應,是一種由大量分子組成的複雜過程,此過程相關的少量基因顯然有所不足,因此為充分運用基因體中所隱含的生物資訊,全基因體定序(whole-genome sequencing)便成為現代分子生物學家的共同目標。
身處大量 DNA 定序數據的時代
DNA 定序儀能產生非常大量的數據,以人工方式檢查長達百萬鹼基對的序列數據,既不實際又缺乏效率,因此,學者開發出能處理這類定序資料的電腦計算方法。自 1980 年代起,多個國際 DNA資料庫便開始儲存上百萬鹼基對的 DNA 數據;到 21 世紀初,數據量便迅速增長至超過 10 億鹼基對。2022 年,總鹼基對數量達到 3 萬 4 千兆,而這就是現在處理的數量。有鑑於此,團隊必須運用資料科學的技術,協助處理如此巨大的資料集。為破解序列資料中「編碼」的分子生物資訊,第一個問題就是如何開發能處理大量序列的分析方法。事實上,隨著 DNA 定序技術的發展,許多用於定序分析的多功能軟體工具也陸續問世。
開發基因體瀏覽工具
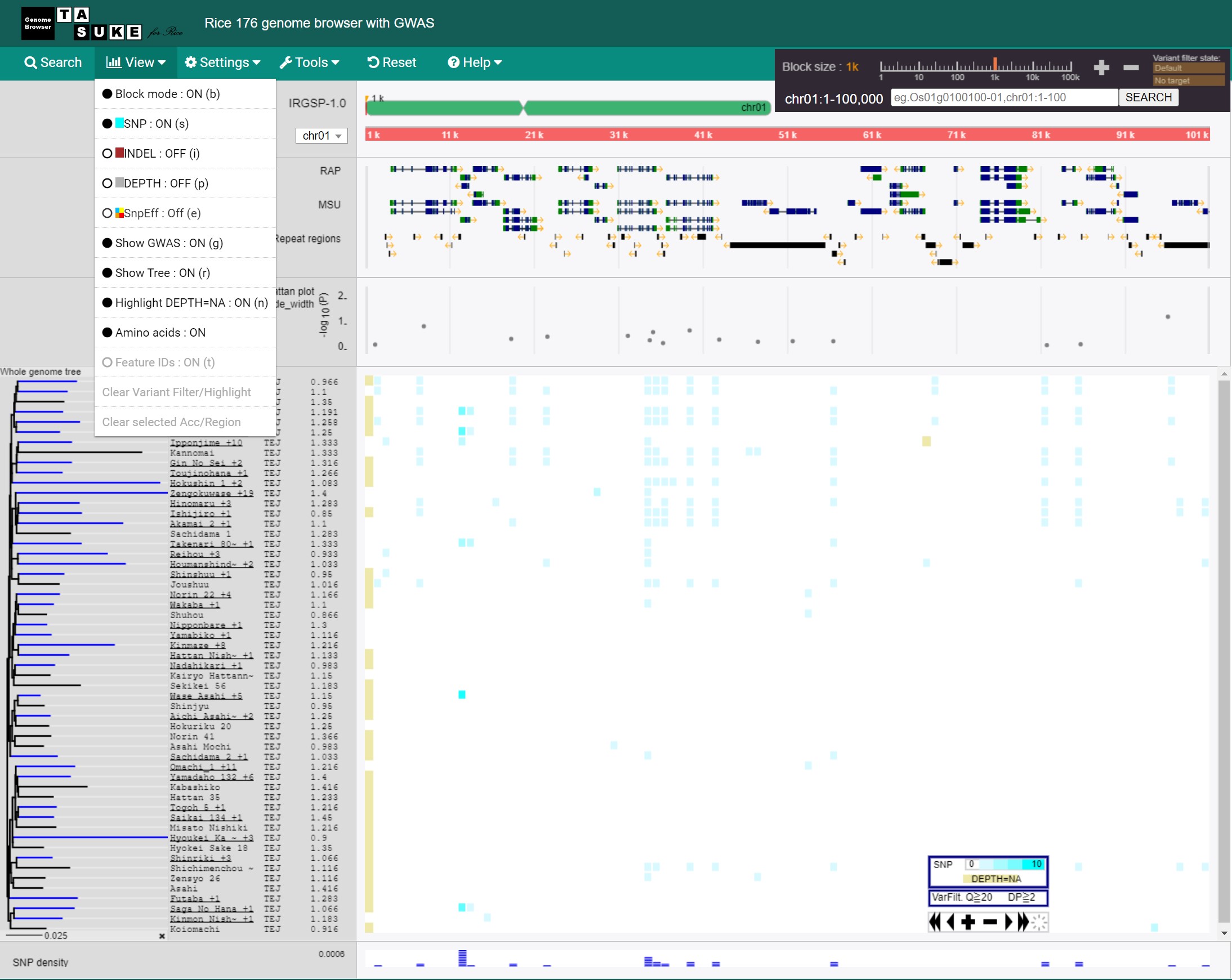
另外值得注意的是,雖然具有分析功能的電腦程式能便利的概述大資料集,研究團隊卻通常必須檢查整組數據。另外, 鑑於某些技術因素,如今的定序儀通常會產生數量可觀且高度破碎的 DNA 序列。因此,了解基因體中有哪些已經定序或尚未定序的部分便十分重要。基因體中微小的不同之處稱為單核苷酸多型性(single-nucleotide polymorphism,簡稱SNP),可以透過比較研究進行偵測。研究人員通常會想評估 SNP 的位置是否在某基因體中有所偏差、哪些基因的位置與 SNP 的位置有所重疊等,這些都是研究者想以視覺化的方式檢測數據的原因,有鑑於此,全球農業科技與基因體科學碩士學位學程教授伊藤剛及其團隊為了達成此目標開發了「TASUKE+」。TASUKE+能將上百組基因體定序的資料集(https://tasuke.dna.affrc.go.jp/)予以視覺化,SNP 的數量將以顏色量表的方式呈現,且 SNP 會與已註解的基因平行顯示,並以樹狀結構呈現樣本之間的關係。TASUKE+也有其他功能,能供此領域的專家使用,以取得更深入的基因體分析資訊。基因定序技術將來必定會大幅進步,而伊藤剛教授正帶領其臺大研究團隊進一步改良TASUKE+,用以處理最新且不同類型的全基因體定序資料。
如需瀏覽近期的會議簡報,請參閱下列 URL:https://plan.core-apps.com/pag_2023/abstract/963d7d6c9252da3647857a6b5d06c83